基于Red同源重组法的大肠杆菌基因组编辑
大肠杆菌基因组编辑的传统方法是利用自身的Red系统对外源进入的DNA进行同源重组,从而实现目标基因的等位替换。通过设计靶基因的同源融合片段,将其克隆至自杀载体中,自杀载体通过接合输入到靶细菌。通过抗生素筛选细菌基因组靶位点整合有自杀载体的插入突变株。在第二轮反向选择压力下,只有大肠杆菌基因组发生第二次同源重组并丢失自杀质粒才可以存活。
Red/ET 同源重组技术(Red/ET recombineering)是由来源于大肠杆菌 λ 噬菌体的蛋白对 Redα/Redβ 或来源于 Rac 原噬菌体的蛋白对 RecE/RecT 所介导的基于短同源臂(40–50 bp)的同源重组技术,能对宿 主 DNA 序列进行快速、高效、精确的修饰和操作。
1998 年,Zhang 等发现大肠杆菌(Escherichia coli) Rac 原噬菌体上的操纵子 recE/recT 编码的 蛋白能够高效地介导体内同源重组的发生[1],而且 Muyers 等发现来源于大肠杆菌 λ 噬菌体的 Redα/ Redβ 蛋白与 RecE/RecT 蛋白有类似的介导同源重 组的功能[2],Stewart 等将 Redα/Redβ 和 RecE/RecT 重组酶体系合并命名为“Red/ET 重组工程(Red/ET recombineering)”[3]。与体内自然发生的同源重组相 比,Red/ET 重组系统只需要 40–50 bp 的同源臂就能 实现高效的同源重组,这一点突破了酶切位点和长 片段 PCR 扩增的局限性对传统基因操作技术的限 制,通过合成引物的方式将短同源臂加在 PCR 产物 的两端,可以与任意位置的目标序列进行同源重组,同时 Red/ET 同源重组技术的操作过程在大肠杆菌 中完成,极大缩短了构建载体和基因修饰的步骤与 时间,因此 Red/ET 同源重组技术的应用已经越来越 广泛。
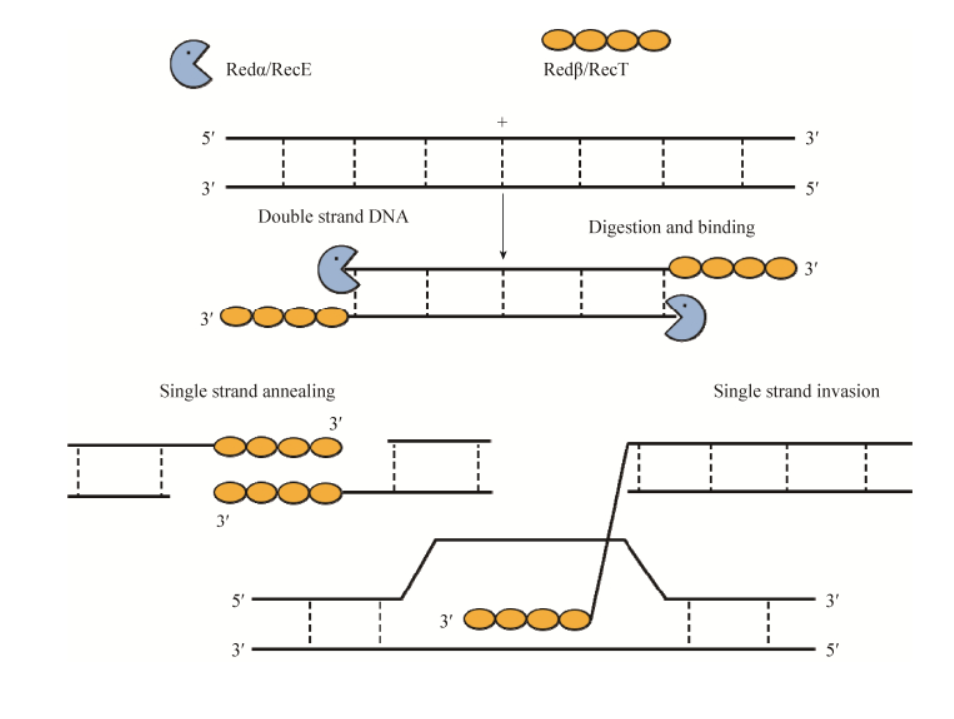
Red/ET 作用机制示意图
Red/ET 重组工程结合反向筛选技术,可以对 DNA 进行无痕敲除、定点突变和模块替换等修饰。以噬菌体蛋白为基础构建的同源重组系统在 细菌基因修饰中应用广泛,不仅可以在大肠杆菌 中高效地修饰 DNA 序列,同样可以在其他多种细 菌中使用(表 1)。
来自大肠杆菌噬菌体的 Red/ET 重组系统只 能在部分与大肠杆菌亲缘关系较近的革兰氏阴性 菌中应用,而在与其亲缘关系较远的菌类中,该 系统的功能是受限制的。然而 Red/ET 重组系统 的建立却为我们在其他细菌基因组或者其噬菌 体基因组中寻找类似的重组酶蛋白提供了理论依 据和技术指导,目前已有多种利用细菌自身噬菌 体或原噬菌体的重组蛋白建立起的具有种属特 异性同源重组工程系统。2007 年 Kessel 等采用来 自革兰氏阳性细菌耻垢分枝杆菌(Mycobacterium smegmatis)噬菌体 Che9c 中的 Redα/β 类似蛋白 gp60/61,构建了分枝杆菌特异性的重组工程系统, 能够以带有 50 bp 短同源臂的单链寡核苷酸作为底 物进行同源重组,通过点突变失活质粒上的潮霉素 抗性基因,在耻垢分枝杆菌和结核分枝杆菌中的重 组效率分别可达到 104 CFU/μg DNA 和 103 CFU/μg DNA,而来源于大肠杆菌的 RecET 和 Redαβγ 则不 能在分枝杆菌中有效介导单链重组[24–25]。2012 年 Pijkeren 等在罗伊氏乳杆菌(Lactobacillus reuteri)中 找到与 recT 同源的 2 个基因 recT1 和 recT2,2 个基因编码的蛋白能够在乳酸杆菌中有效地介导 单链重组的发生[26]。
Red/ET 同源重组技术还能免建库从基因组中 直接克隆大型基因簇(图 3-B,表 2),大大促进了微 生物次级代谢产物的异源表达研究。
Red/ET 同源重组技术不仅可以对大肠杆菌中 DNA 分子的任意位置进行精确的修饰,包括单碱 基改变和基因的插入与删除,而且现在已经能够 在多个其他细菌中工作。将来继续发现新的宿主 特异性重组酶,并使之能够在更多的细菌中(假单 胞菌、芽孢杆菌、伯克氏菌等)建立以 Red/ET 同 源重组技术为基础的高效遗传操作体系,促进微 生物功能基因组和基因组挖掘的发展。以全长 RecET 介导的线线重组为基础的免建库直接克隆 技术为大片段基因簇的研究奠定了基础,极大促 进了后基因组时代以异源表达为策略的微生物次 级代谢产物的发现和开发。 随着分子生物学和基因工程技术的发展, Red/ET 同源重组技术也在不断发展和完善,已经 成为基因功能研究中一项热门的技术,在基因组 学研究和微生物基因组功能挖掘中将扮演更重要的角色。
参考文献:
[1] Zhang YM, Buchholz F, Muyrers JPP, Stewart AF. A new logic for DNA engineering using recombination in Escherichia coli. Nature Genetics, 1998, 20(2): 123–128.
[2] Muyrers JPP, Zhang YM, Testa G, Stewart AF. Rapid modification of bacterial artificial chromosomes by ET-recombination. Nucleic Acids Research, 1999, 27(6): 1555–1557.
[3] Zhang YM, Muyrers JPP, Rientjes J, Stewart AF. Phage annealing proteins promote oligonucleotide-directed mutagenesis in Escherichia coli and mouse ES cells. BMC Molecular Biology, 2003, 4(1): 1.
[4] Muyrers JPP, Zhang YM, Stewart AF. Techniques: recombinogenic engineering-new options for cloning and manipulating DNA. Trends in Biochemical Sciences, 2001, 26(5): 325–331.
[5] Sharan SK, Thomason LC, Kuznetsov SG, Court DL. Recombineering: a homologous recombination-based method of genetic engineering. Nature Protocols, 2009, 4(2): 206–223.
[6] Little JW. An exonuclease induced by bacteriophage λ II. Nature of the enzymatic reaction. Journal of Biological Chemistry, 1967, 242(4): 679–686.
[7] Karakousis G, Ye N, Li Z, Chiu SK, Reddy G, Radding CM. The beta protein of phage λ binds preferentially to an intermediate in DNA renaturation. Journal of Molecular Biology, 1998, 276(4): 721–731.
[8] Li Z, Karakousis G, Chiu SK, Reddy G, Radding CM. The beta protein of phage λ promotes strand exchange. Journal of Molecular Biology, 1998, 276(4): 733–744.
[9] Fu J, Bian XY, Hu S, Wang HL, Huang F, Seibert PM, Plaza A, Xia LQ, Müller R, Stewart AF, Zhang YM. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nature Biotechnology, 2012, 30(5): 440–446.
[10] Cobb RE, Zhao HM. Direct cloning of large genomic sequences. Nature Biotechnology, 2012, 30(5): 405–406.
[11] Nawy T. Molecular biology: capturing sequences for bioprospecting. Nature Methods, 2012, 9(6): 532.
[12] Wang HH, Isaacs FJ, Carr PA, Sun ZZ, Xu G, Forest CR, Church GM. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894–898.
[13] Isaacs FJ, Carr PA, Wang HH, Lajoie MJ, Sterling B, Kraal L, Tolonen AC, Gianoulis TA, Goodman DB, Reppas NB, Emig CJ, Bang D, Hwang SJ, Jewett MC, Jacobson JM, Church GM. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333(6040): 348–353. [14] Wang HH, Kim H, Cong L, Jeong J, Bang D, Church GM. Genome-scale promoter engineering by coselection MAGE. Nature Methods, 2012, 9(6): 591–593.